近日,瓯江实验室帅建伟团队在期刊Advanced Science上发表题为“A Multi-Task Self-Supervised Strategy for Predicting Molecular Properties and FGFR1 Inhibitors”的研究论文。
生物医学研究和技术虽然取得了显著进展,药物的发现和开发仍然是一项复杂而充满挑战性的任务。这种复杂性源于药物研发过程中各个阶段所特有的挑战和困难。在药物发现阶段,需要进行广泛的基础科学研究,包括深入理解疾病机制、识别潜在的药物靶点、筛选和优化化合物等。为了筛选评估每种化合物对所有可能的蛋白质靶标的影响,传统的实验方法往往需要耗费大量的时间和资源。为了突破这些限制,研究人员越来越多地采用计算方法来加速药物发现。其中,深度学习方法作为一种高效的技术手段,代表了分子表示学习领域的重大突破。

该研究通过利用大规模无标记化合物数据学习,自监督深度学习模型能够学习到更丰富、更有效的分子表示,从而显著提高下游预测任务的性能。该研究显示,多任务预训练框架在涵盖各种药物发现任务的基准生物医学数据集上表现出了优越的性能。值得注意的是,在识别FGFR1的潜在抑制剂这个重要的实际应用方面,所提出的模型能够更高效、更准确地预测靶点的潜在抑制剂。此外,他们还通过分子对接和分子动力学模拟,进一步证实了预测结果的准确性。该研究展现了深度学习算法在加速药物发现和提供对分子相互作用的见解方面的巨大潜力。
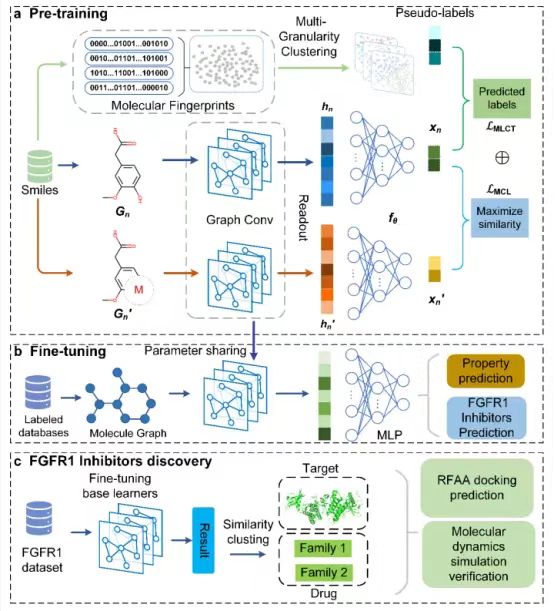
多任务预训练模型药物发现实验流程:(a)预训练框架。首先,数据增强技术应用于原始输入数据,以创建各种增强版本。然后将这些分子图数据输入特征提取模块以提取潜在特征。最后,将增广后的数据用于两个学习任务。(b)通过迁移学习针对下游预测任务微调。(c)预测FGFR1潜在的抑制剂并通过分子对接和分子动力学验证预测结果。
瓯江实验室帅建伟研究员和辽宁科技大学赵琪教授为本文共同通讯作者,辽宁科技大学2022级硕士研究生杨鑫和帅建伟研究员团队的王洋副研究员为本文共同第一作者。
原文链接
https://doi.org/10.1002/advs.202412987
来源:帅建伟课题组
编辑:冯如如
排版:吕旭东
审核:凌树宽
-
Nat Commun 丨瓯江实验室李校堃院士团队肖健课题组开发生长因子动态相位自适应水凝胶实现耐药菌感染创面修复
原文地址:/scientific/912.html -
Nat Microbio | 瓯江实验室苏建忠团队开发单细胞水平遗传算法揭示宿主遗传-菌群-细胞类型内在关联性
原文地址:http://www.ojlab.cn/keyanjinzhan/901.html -
Science Translational Medicine | 突破争议!瓯江实验室苏建忠/瞿佳团队发现人类视网膜再生"种子细胞", 数亿致盲患者迎来曙光
原文地址:/keyanjinzhan/850.html -
EMBO Molecular Medicine | 瓯江实验室王旭课题组发现OTUB2是缺血性脑卒中的重要治疗靶点
原文地址:http://www.ojlab.cn/keyanjinzhan/809.html



